8月22日,集美大学水产学院王志勇教授团队在Advanced Science(2025年 IF=14.1,5年IF=15.6)杂志上发表了题为《T2T Genomes Unveil Centromere Architecture and Adaptive Divergence in Large Yellow Croaker (Larimichthys crocea)》的研究论文,该研究提供了迄今为止最完整的闽-粤东族和岱衢族大黄鱼参考基因组;同时,结合多组学数据系统解析了着丝粒特征及5S rRNA分布模式,并开展了不同种群大黄鱼适应性演化分析。该研究为大黄鱼重要经济性状遗传机制解析和遗传改良工作的开展提供了重要基础。全文链接:https://advanced.onlinelibrary.wiley.com/doi/10.1002/advs.202506374.

摘要:
大黄鱼作为重要的海水养殖鱼类,高质量基因组的缺失制约着其着丝粒和适应性进化研究。本研究首次公布了闽-粤东族和岱衢族大黄鱼端粒到端粒(T2T)无间隙基因组,这两个种群于418万年前发生分化。大黄鱼着丝粒非编码区域由Cen-42特征序列及LTR/ERV1转座元件入侵构成,编码区域在两个种群中表现出种类和数量的差异,这可能是由群体快速扩张和选择压力共同驱动。此外,5S rRNA在超过10条染色体的短臂区域形成超大型串联重复簇。T2T基因组的组装解决了既往版本中未组装的区域,在闽粤东族和岱衢族中分别多注释到533、351个蛋白编码基因。鉴定出了4个闽粤东族特异的结构变异,分别位于pla2g4a、eno2、ptprb和itrp3基因上。比较基因组分析显示两个种群在代谢效率、花生稀稀酸代谢、化学感知和昼夜节律等方面表现出差异化适应特征。这些发现深化了对大黄鱼进化机制的理解,也为其资源保护和遗传育种提供了宝贵资源。
结果:
(1)成功将闽粤东族和岱衢族大黄鱼基因组24条染色体均提升至T2T水平,两族基因组大小分别为708.23和706.61Mb。与已发表的大黄鱼基因组相比,新注释到了533、351个蛋白编码基因。
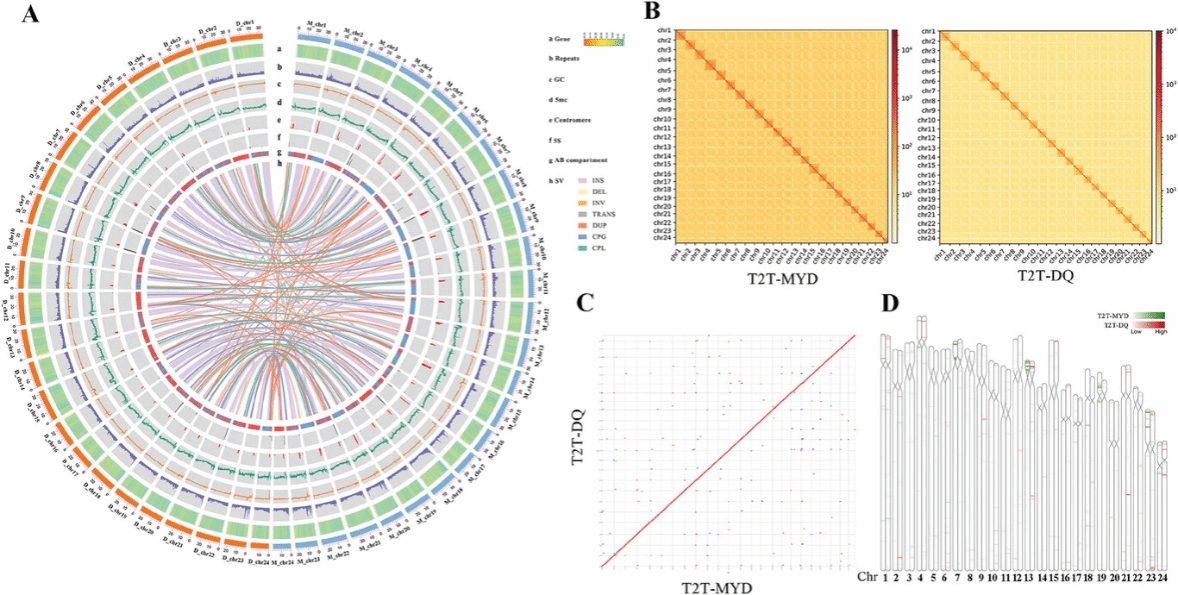
(2)系统解析了大黄鱼着丝粒序列特征:着丝粒区域由Cen-42重复序列和LTR/ERV1入侵构成,具有转录活性,具有较高的5mc5mC甲基化频率和较低的H3K4me3信号。两个种群着丝粒区域分化程度较高,可能受到群体大小波动及选择压力的共同影响。
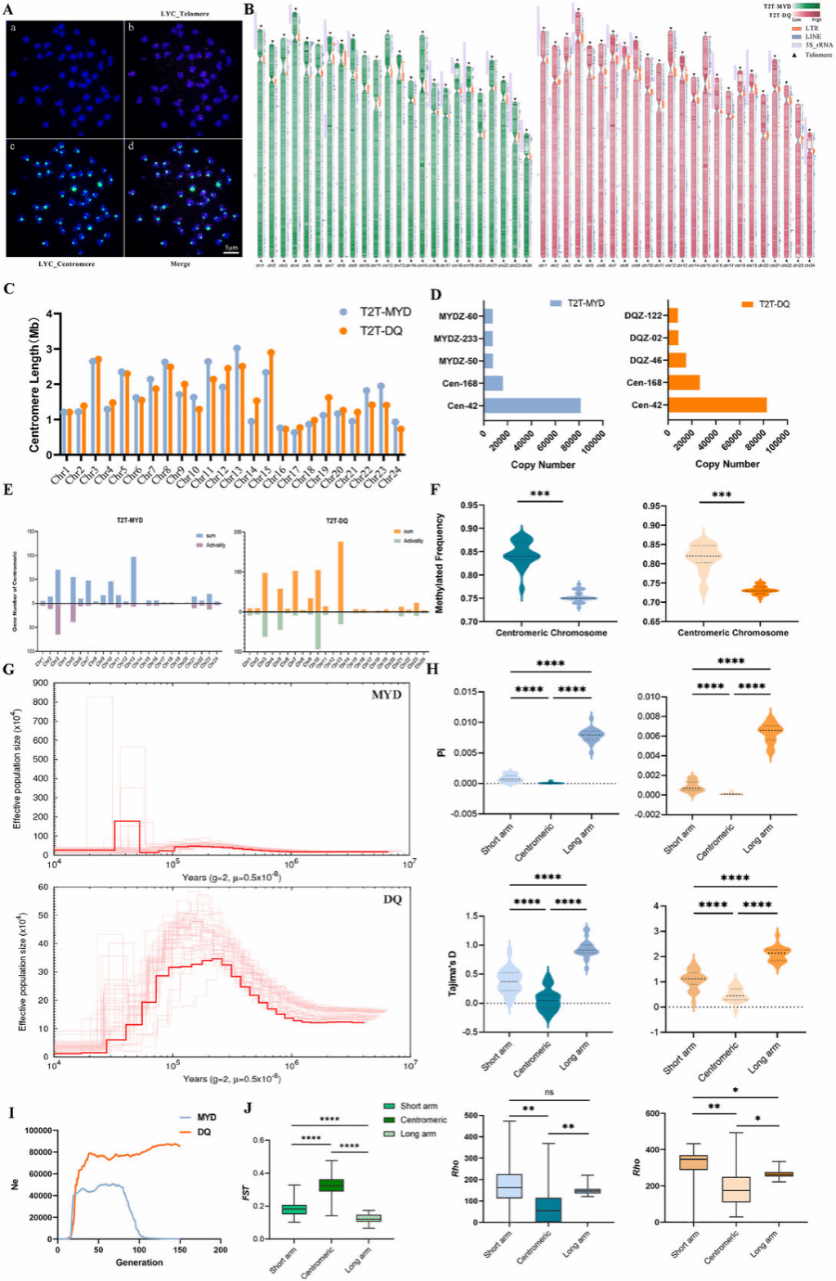
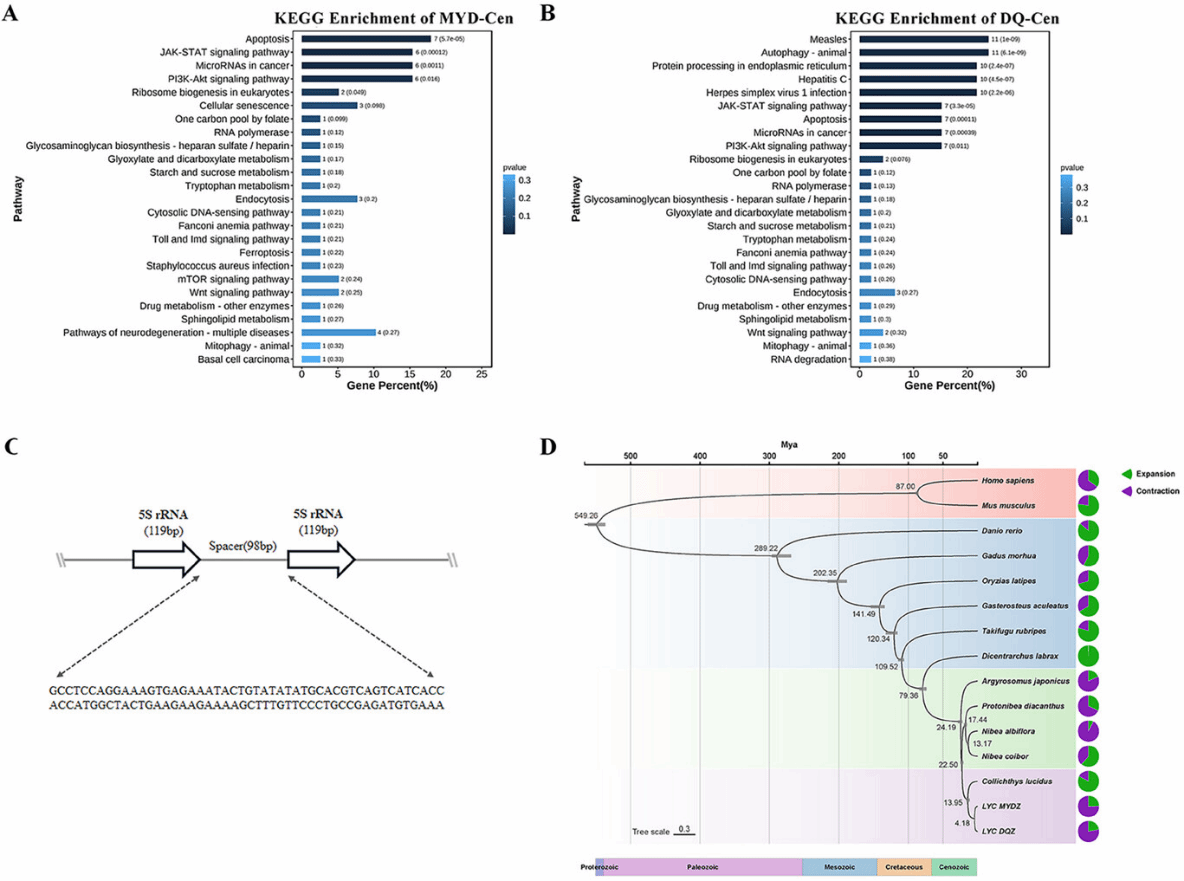
(3)5S rRNA在大黄鱼基因组中呈弥散分布,与一个98bp的重复序列交替串联分布,且与LINE/L2共定位,这个模式可能是大黄鱼特有的。此外,进化分析显示两个种群在约418万年前开始分化,基因家族和选择压力分析显示两个种群在代谢效率、花生稀稀酸代谢、化学感知和昼夜节律等方面表现出差异,并在碱基水平和表达水平进行了验证。
集美大学水产学院博士研究生崔瑜为该论文第一作者,参与该工作的还有博士生原应博,硕士生王皕、吴宝兰,鱼类遗传育种研究团队蔡明夷教授、方铭教授、韩芳教授,以及宁波市海洋与渔业研究院的沈伟良和吴雄飞研究员;集美大学水产学院的王志勇教授为通讯作者。